团队在佐治亚理工学院采用深度学习对scRNA-seq数据进行批次效应和条件效应建模
 发布于2024-12-15 阅读(0)
发布于2024-12-15 阅读(0)
扫一扫,手机访问

编辑 | 萝卜皮
单细胞RNA测序(scRNA-seq)已成为广泛应用于疾病研究的重要工具。在这些研究中,样本批次的收集涵盖了不同条件,包括人口群体、疾病阶段和药物治疗。需要注意的是,样本批次之间的差异往往是由批次效应和条件效应的结合引起的。批次效应主要是技术混杂因素带来的差异,而条件效应则是由生物变异引起的。这种混合体的存在使得研究人员需要仔细考虑如何正确解释和处理数据,以确保结果的准确性和可靠性。
然而,目前的批次效应消除方法通常会同时除去技术批次效应和有意义的条件效应,而干扰预测方法则只专注于条件效应。这导致了基因表达预测的不准确,因为未考虑到批次效应的影响。
最新的研究发现,佐治亚理工学院(Georgia Institute of Technology,GT)的科研团队开发了一种名为scDisInFact的深度学习框架。该框架专门用于对单细胞RNA测序(scRNA-seq)数据中的批次效应和条件效应进行建模。这项研究的意义在于帮助科学家更好地理解单细胞水平的基因表达变化,从而推动生物医学领域的进步。通过scDisInFact,研究人员可以更准确地识别和分析数据中的影响因素,为细胞组学研究提供更深入的
scDisInFact致力于研究将条件效应与批次效应区分开来的潜在因素,以便能够同时处理批次效应的消除、条件相关关键基因的检测以及扰动的预测。这个工作的重点在于有效地应对数据中的变异性,以便更准确地识别和解释基因表达数据中的差异。通过这种方法,可以更好地理解基因表达数据背后的生物学含义,为进一步的
研究团队对scDisInFact在模拟和真实数据集上的表现进行了评估,并与各项任务的基准方法进行了比较。研究结果显示,scDisInFact的性能优于目前仅专注于单一任务的方法,为整合和预测多批次、多条件的单细胞RNA测序数据提供了更为全面和准确的解决方案。
该研究以「scDisInFact: disentangled learning for integration and prediction of multi-batch multi-condition single-cell RNA-sequencing data」为题,于 2024 年 1 月 30 日发布在《Nature Communications》。

单细胞RNA测序(scRNA-seq)是一种能够测量每个细胞中基因表达水平的技术。它已被广泛运用于疾病研究,可以通过收集来自不同疾病阶段或接受不同药物治疗的捐赠者的样本来进行分析。这项技术的发展为研究人员提供了一种更深入了解细胞在不同条件下如何表达基因的方式,有助于揭示疾病的发病机制和药物治疗的效果。通过单细
因此,每个样本的 scRNA-seq 计数矩阵与供体的一种或多种生物学状况相关,这些生物学状况可以是年龄、性别、药物治疗、疾病严重程度等。同时,研究同一疾病的数据集通常是在不同批次中获得的,这会引入跨批次的技术差异(也称为批次效应)。
实际上,疾病研究数据集中的可用样本可能来自不同的条件和批次。研究人员将此类数据集称为多批次多条件数据集。在此类数据集中,同一批次生成的不同生物条件的数据矩阵之间存在由条件效应引起的生物变异,而同一条件但不同批次的数据矩阵之间存在批次效应引起的技术变异。
因此,这些数据矩阵之间的差异是批次效应(技术变异)和条件效应(生物变异)的混合体,这使得充分利用这些数据集潜力的过程变得复杂。
科学家考虑了使用多批次多条件数据集进行疾病研究时需要解决的一些计算挑战:(1)消除批次效应,同时保留生物条件效应;(2)检测与生物状况相关的关键基因;(3)预测对应于一定条件的未见数据矩阵,也称为扰动预测任务。学界已经针对每个问题分别设计了方法,但没有现有的方法可以共同解决这三个问题。
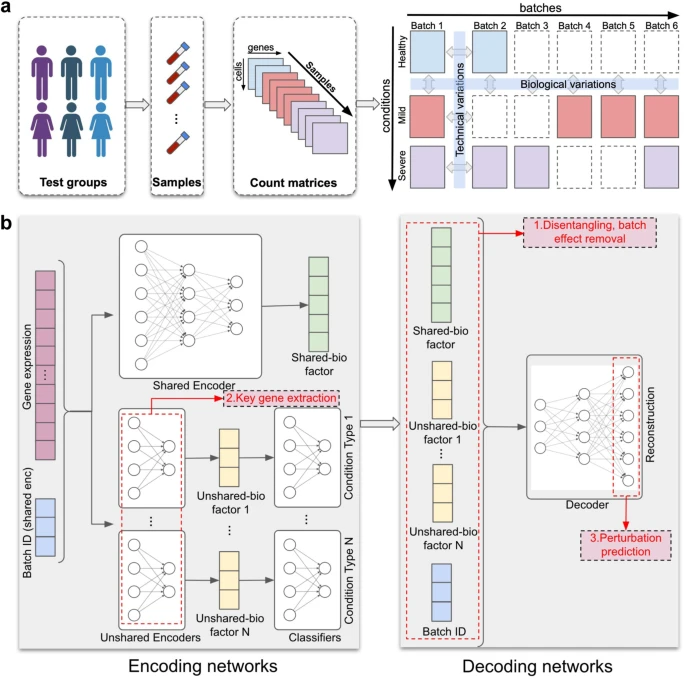
在最新的研究中,佐治亚理工学院的研究团队提出了 scDisInFact(single cell disentangled Integration preserving condition-specific Factors),这是第一个可以执行所有三个任务的方法:批次效应消除、条件相关关键基因(CKG)检测以及多批次多条件 scRNA-seq 数据集上的扰动预测。
scDisInFact 是基于解纠缠分自动编码器框架设计的。它将多批次多条件数据集中的变化分解为编码所有数据矩阵共享的生物信息、特定条件的生物信息和技术批次效应的潜在因素。解开的潜在空间允许 scDisInFact 执行另外两项任务,即 CKG 检测和扰动预测,并克服每个任务现有方法的限制。
特别是,解开的因子允许 scDisInFact 消除批次效应,同时保留基因表达数据中的条件效应。此外,scDisInFact 扩展了现有扰动预测方法的多功能性,因为 (1) 它可以对多种条件类型的影响进行建模,(2) 它可以跨数据集中的条件和批次的任意组合进行数据预测。
研究人员在消除批次效应和 CKG 检测方面比较了 scDisInFact 和 scINSIGHT(一种同类型方法,使用非负矩阵分解将 scRNA-seq 矩阵分解为通用模块和条件特定模块。)。由于 scINSIGHT 不进行扰动预测,因此该团队在扰动预测方面将 scDisInFact 与 scGen 和 scPreGAN 进行了比较。
对于消除批次效应,scDisInFact 仅消除批次效应并保留数据矩阵之间的生物学差异。对于与病情相关的关键基因检测,scDisInFact不仅可以高水平输出CKG,而且扰动预测结果还可以用于寻找在特定细胞或细胞类型中从一种条件组合到任何其他条件组合差异表达的基因。
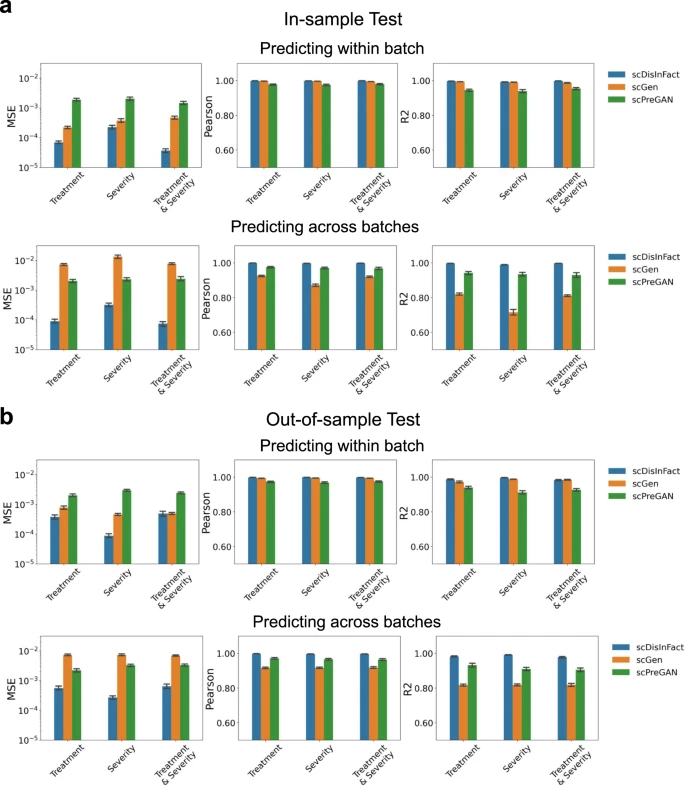
图示:模拟数据集上的扰动预测结果。(来源:论文)
对于扰动预测,scDisInFact 对与捐赠者相关的多种条件类型进行建模,并可以预测从条件组合到所研究的任何其他组合的数据。这使得在复杂场景中的应用成为可能,例如预测多种药物组合的效果。

图示:模拟数据集的结果。(来源:论文)
该团队在模拟和真实数据集上测试了 scDisInFact,发现它在各种任务中都优于基线方法。由于其卓越的性能和多任务能力,scDisInFact 可用于全面分析多批次多条件 scRNA-seq 数据集,有助于更深入地了解疾病进展和患者对药物治疗的反应。
论文链接:https://www.nature.com/articles/s41467-024-45227-w
上一篇:扩展两台笔记本的屏幕
产品推荐
-

售后无忧
立即购买>- DAEMON Tools Lite 10【序列号终身授权 + 中文版 + Win】
-
¥150.00
office旗舰店
-

售后无忧
立即购买>- DAEMON Tools Ultra 5【序列号终身授权 + 中文版 + Win】
-
¥198.00
office旗舰店
-

售后无忧
立即购买>- DAEMON Tools Pro 8【序列号终身授权 + 中文版 + Win】
-
¥189.00
office旗舰店
-

售后无忧
立即购买>- CorelDRAW X8 简体中文【标准版 + Win】
-
¥1788.00
office旗舰店
-
 正版软件
正版软件
- trc20和erc20哪个更快
- TRC-20比ERC-20更快,因为它具有更短的区块时间和更少的网络拥塞。TRC-20每秒可处理2,000笔交易,而ERC-20为15-25笔;TRC-20区块时间为3秒,而ERC-20为12-15秒。此外,Tron网络通常不太拥挤,节点数量、网络稳定性和交易费用也更具优势。
- 18分钟前 0
-
 正版软件
正版软件
- 人人都可成为AI开发者!李彦宏带来三大AI开发神器
- 2023年初崛起为科技领域最亮的星,展示了凯AI技术变革经济社会的巨大潜能。经过一年多的技术突破和市场验证,进入2024年,大模型的巨大价值已经得到主流国家的共识,主流科技公司纷纷布局。美国、中国等国家,正处于引领大模型产业发展的未来。在国内科技公司中,率先发布文心大模型的百度,已经取得了不凡成果。在今日举办的Create2024百度AI开发者大会上,百度创始人、董事长兼首席执行官李彦宏在主题演讲中透露,文心一言用户数突破2亿,文心大模型成为中国最领先、应用最广泛的AI基础模型。与此同时,李彦宏还打算让所
- 43分钟前 大模型 AI开发 0
-
 正版软件
正版软件
- AI失业潮已来,程序员职业是否真的会消失?
- 最近,有消息称OpenAI将发布突破性的多模态机器学习模型GPT-4o。关于人工智能引发全球性失业潮的报道再次占据各大媒体头条。国际货币基金组织总经理克里斯塔利娜·格奥尔基耶娃博士警告称,随着企业大量采用人工智能技术,全球劳动力市场将遭遇“海啸”,人工智能可能会消灭全球近一半(40%)的工作岗位以及美国和英国等发达经济体一半以上(60%)的工作岗位。克里斯塔利娜·格奥尔基耶娃博士在一次演讲中指出,人工智能的发展将对各行各业产生深远影响。尽管人工智能有助于提高生产效率和创新能力,但也将导致大规模的岗位消失。
- 58分钟前 人工智能 机器学习 OpenAI 0
-
正版软件
- 虚拟货币交易平台前十 全球最大的虚拟货币交易所排行
- 全球虚拟货币交易所排名根据交易量和用户数量,排名如下:交易量:币安火币OKXFTX币安美国用户数量:币安火币CoinbaseFTXOKX
- 1小时前 02:34 0
-
正版软件
- 中国正规数字货币交易平台
- 中国境内仅有7家正规数字货币交易平台:火币网、OKX、币安中国、抹茶网、库币网、MXC抹茶、BitMart币玛特。这些平台已获得境外金融牌照,可为中国用户提供数字货币交易服务,但不得从事法币交易或代理其他交易平台。
- 1小时前 02:14 0
最新发布
-
 1
1
- 阿里追捧的中台,“热度”退了?
- 1983天前
-
 2
2
- Overture设置踏板标记的方法
- 1820天前
-
 3
3
- 思杰马克丁取得CleanMyMac中国区独家发行授权
- 1809天前
-
 4
4
- IBM:20万台Mac让公司职工在工作中更快乐 更多产
- 2008天前
-
 5
5
- 报道称微软一直在悄然游说反对“维修权”立法!
- 1974天前
-
 6
6
- 美国怀疑华为窃取商业机密 华为:身正不怕影子斜
- 1970天前
-
 7
7
- 三星被曝正与联发科接洽 A系列手机有望搭载其5G芯片
- 1984天前
-
 8
8
- 环球墨非完成千万级融资 联合企业集团投资
- 2006天前
-
 9
9
相关推荐
热门关注
-

- Xshell 6 简体中文
- ¥899.00-¥1149.00
-

- DaVinci Resolve Studio 16 简体中文
- ¥2550.00-¥2550.00
-

- Camtasia 2019 简体中文
- ¥689.00-¥689.00
-

- Luminar 3 简体中文
- ¥288.00-¥288.00
-

- Apowersoft 录屏王 简体中文
- ¥129.00-¥339.00